Die Robustheit und Regulierbarkeit des pflanzlichen Immunsystem
Forschungsbericht (importiert) 2015 - MPI für Pflanzenzüchtungsforschung
Das Immunsystem der Pflanze
Pflanzen sind kontinuierlich mit zahlreichen Mikroben konfrontiert und obwohl diese potenziell Krankheiten auslösen können, bleiben die meisten Pflanzen aufgrund eines funktionierenden Immunsystems gesund. Pflanzen können nämlich fremde mikrobielle Moleküle erkennen und lösen so eine Reihe von Immunantworten aus. Bakterielles Flagellin wird beispielsweise von einem Rezeptor an der pflanzlichen Zelloberfläche gebunden, wodurch Signalkaskaden innerhalb des Immunsystems aktiviert werden. Mikroben antworten darauf mit Virulenzfaktoren, die im Verlauf der Ko-Evolution von Mikroorganismen und ihren pflanzlichen Wirten entstanden sind, Teile dieser Immunantwort unterdrücken und somit ein Leben innerhalb der Pflanze ermöglichen. Als Gegenmaßnahme haben manche Pflanzenspezies Rezeptoren entwickelt, die die direkte oder indirekte Erkennung mikrobieller Virulenzfaktoren ermöglichen [1].
In Anbetracht der deutlich längeren Generationszyklen von Pflanzen im Vergleich zu Mikroben kann die Entstehung solcher Gegenmaßnamen nicht immer mit der Evolution neuer Virulenzfaktoren mithalten. Deshalb muss das pflanzeneigene Immunsystem robust gegen jede Art mikrobieller Störungen sein, um sich prinzipiell vor einer Infektion durch Pathogene zu schützen. Wie aber kann ein solches System robust gegen äußere Störungen sein? Man könnte annehmen, dass eine konstitutive Aktivierung vieler verschiedener Immunantworten reichen würde, da es für Mikroben unmöglich sein sollte, alle Immunantworten gleichzeitig zu stören. Das aber wäre aufgrund von trade-offs zwischen Immunität und anderen physiologischen Prozessen keine Option für die Pflanze (Abb. 1; [2]).
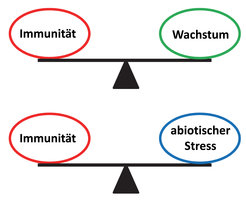
Abb. 1: Trade-offs zwischen Immunität und ausgewählten physiologischen Prozessen. Nur eine Balance garantiert, dass die Abwehr gegen Pathogene sich nicht negativ beispielsweise auf Wachstum oder die Entgegnung auf abiotischen Stress wie zum Beispiel Kälte oder starkes Sonnenlicht auswirkt.
Eine zu starke Immunantwort reduziert zum Beispiel die Fitness der Pflanze, indem sie das pflanzliche Wachstum unterdrückt. Es muss daher gezielt reguliert werden, um dessen Aktivierung nur bei Bedarf und in angebrachter Stärke zu gewährleisten. Zusätzlich muss das Immunsystem Robustheit vorweisen. Damit stellt sich die Frage, wie innerhalb pflanzlicher Immunsignalnetzwerke Robustheit und Regulierbarkeit vereint werden können – nicht zuletzt auch deswegen, weil Pflanzenpathogene den Ertrag und die Qualität von Nutzpflanzen empfindlich reduzieren und wirtschaftlichen Schaden anrichten können. Ein Ziel der Erforschung des pflanzlichen Immunsystems ist daher besonders, neue Möglichkeiten für die Produktion Pathogen resistenter Nutzpflanzen zu finden.
Komplexe Phytohormon-Signalnetzwerke im Immunsystem der Pflanze
Pflanzen produzieren eine Vielzahl an Signalmolekülen, darunter Phytohormone, die neben verschiedensten physiologischen Prozessen auch an der Regulierung des Immunsystems beteiligt sind [3]. Zum Beispiel ist Salicylsäure (SA) zu großen Teilen an der Aktivierung der Immunantwort gegen biotrophe Pathogene beteiligt - Pathogene, die die Pflanze nach erfolgreicher Infektion am Leben halten. Jasmonsäure (JA) hingegen induziert die Immunantwort gegen Pflanzenfresser und nekrotrophe Pathogene, die die Pflanze töten und sich nachfolgend von dem abgestorbenen Material ernähren. Die Signalwege, die durch diese beiden Phytohormone reguliert werden, sind eng miteinander vernetzt und wirken generell antagonistisch aufeinander. Man nimmt an, dass bei Aktivierung des einen Signalweges der andere Signalweg unterdrückt wird. Studien an der Modellpflanze Arabidopsis thaliana (Acker-Schmalwand) haben gezeigt, dass tatsächlich eine Induzierung des SA-abhängigen Signalweges die Aktivierung JA-abhängiger Signalwege reduziert und umgekehrt. Diese Beobachtungen berufen sich entweder auf einfache genetische Untersuchungen von Mutanten, bei denen einzelne Signalwege ausgeschaltet wurden, oder auf Versuche mit direktem Einsatz der jeweiligen Phytohormone. Analysen mithilfe von systembiologischen Ansätzen, kombiniert mit computerbasierten Modellierungen, konnten jedoch deutlich komplexere Eigenschaften der Interaktion von SA- und JA-Signalwegen aufdecken. So zeigte sich beispielsweise, dass beide Signalwege kompensatorisch miteinander in Beziehung treten [4-7].
In A. thaliana hat das Gen PAD4 einen positiven Einfluss auf die Akkumulation von SA. Dass pad4-defiziente Mutanten entsprechend anfällig für viele biothrophe Pathogene sind, unterstreicht die Relevanz von PAD4 während der Immunantwort. Im Vergleich zum Wildtyp akkumulieren pad4-Mutanten weniger und JA-defiziente Mutanten mehr SA. Interessant wiederum ist, dass eine Doppelmutante, defizient sowohl in PAD4 als auch in JA, überhaupt kein SA mehr als Antwort auf Flagellin produzieren kann. Analysen mit den jeweiligen Einzelmutanten führten zu der Annahme, dass PAD4 ein positiver und JA ein negativer Regulator der Akkumulation von SA sei. Dieses einfache Modell konnte jedoch nicht das Ergebnis der Doppelmutante erklären, bei der JA ebenfalls positiv auf die Akkumulation von SA wirkt. Wie können diese Beobachtungen also erklärt werden?
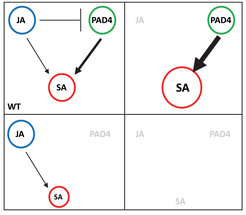
Abb. 2: Ein Netzwerkmodell zweier Signalwege – Jasmonat (JA) und Salicylat (SA) - das Robustheit des pflanzlichen Immunsystems und zugleich dessen Regulierbarkeit ermöglicht. WT, Wildtyp. Nähere Erläuterung im Text.
Abbildung 2 zeigt ein Modell, das die Ergebnisse vollständig vereint: Sowohl JA als auch PAD4 tragen positiv zur Akkumulation von SA bei, wobei PAD4 einen größeren Anteil daran hat. JA-abhängige Signale haben zudem einen negativen Einfluss auf PAD4. Wenn die JA-abhängigen Signale verschwinden, akkumuliert eine größere Menge an SA in der Pflanze, da der negative Einfluss von JA auf PAD4 unterbunden ist. Wenn aber der PAD4 Signalweg wegfällt, führt lediglich - in geringem Maße - der JA-abhängige Signalweg zur Akkumulation von SA. Beim Verlust von PAD4- und JA-abhängigen Signalwegen kann die Pflanze kein SA mehr akkumulieren. Im intakten Netzwerk, also in Wildtyp Pflanzen, wird folglich der Level an SA durch die Wirkung von JA feinreguliert, um eine eventuelle Überakkumulation von SA zu verhindern. Somit ermöglicht der JA-abhängige Signalweg Robustheit und zugleich Regulierbarkeit im Verlauf der Akkumulation von SA. Diese Analysen unterstreichen die Bedeutung von systembasierten Methoden als Mittel zum Verständnis der Natur komplexer Signalnetzwerke.
Die Komplexität des SA-JA-crosstalks ist nur ein Beispiel. Weitere Phytohormon-Signalwege sind eng miteinander verbunden und bilden somit zusammen mit SA und JA ganze Signalnetzwerke. Zusätzlich interagieren die Phytohormon-Signalwege mit andersartigen Signalwegen [8, 9], die dann zusammen das ganzheitliche Signalnetzwerk der Pflanze ausbilden. Ein solches umfassendes Netzwerk ist nicht statisch, sondern dynamisch in Zeit und Raum. Ein Ziel der Untersuchungen ist es, das Verhalten der Pflanze unter verschiedensten Bedingungen zu prognostizieren.
Evolution und Anpassung des pflanzlichen Immunsystems

Kausalstudien wie die hier erläuterten werden meistens nur mit einer oder einer limitierten Anzahl an pflanzlichen Modellorganismen durchgeführt. Dies führt zu der Ungewissheit, inwieweit sich Erkenntnisse solcher Modellorganismen auf die Verbesserung von Nutzpflanzen, die aus unterschiedlichen Pflanzenfamilien stammen, übertragen lassen. Darüber hinaus ist unklar, wie sich das Immunsystem einer individuellen Pflanze entwickelt und wie es sich an äußere Umwelteinflüsse anpasst. In welchem Umfang sind beispielsweise einzelne Komponenten des pflanzlichen Immunsystems konserviert oder aber diversifiziert? Sind die Strukturen und Dynamiken eines definierten Immunnetzwerks unverändert in verschiedenen Spezies vorzufinden? Die Modellpflanze Arabidopsis thaliana gehört zur Familie der Brassicaceae, zu der viele, inzwischen vollständig sequenzierte Nutzpflanzen wie viele Kohlsorten und Raps gehören. Vertreter dieser Familie sind demnach eng miteinander verwandt und weisen dennoch Anpassung an verschiedene Habitate vor.
Studien an mehreren, nah verwandten Spezies ermöglichen eine genauere Betrachtung evolutiver Prozesse, da sie in sich nahe stehenden Arten in kleinen, übersichtlichen Zeiträumen stattgefunden haben müssen. Basierend auf diesen Vorteil untersuchen die Wissenschaftlerinnen und Wissenschaftler parallel die genetische Architektur sowie Transkriptome und Metabolome in mehreren Brassicaceae-Spezies (Abb. 3).
Diese Analysen werden zu neuen, fundamentalen Erkenntnissen im Bereich der Signalnetzwerke pflanzlicher Immunsysteme führen und bei der Erzeugung besserer Nutzpflanzen helfen.


