Die regulatorische Rolle der TFIIH Proteinkinasen in Pflanzen
Forschungsbericht (importiert) 2012 - MPI für Pflanzenzüchtungsforschung
Wie werden Gene abgelesen?
Die RNA-Polymerase II übersetzt die genetische Information in eine Botenribonukleinsäure (mRNA), aus der die nicht-kodierenden Anteile, die sogenannten Introns, noch vor der Proteinbiosynthese wieder herausgeschnitten werden. Sie produziert auch die Mehrzahl der nicht proteinkodierenden microRNAs und der sogenannten trans-acting silencing RNAs. Diese kleinen Ribonukleinsäuren regulieren die Expression anderer Gene, indem sie deren mRNA zerschneiden oder die entsprechenden Gene auf andere Weise stilllegen.
Jede von der RNA-Polymerase II produzierte Ribonukleinsäure ist durch eine Kappe aus Methyl-Guanosin und einen Schwanz aus Poly-Adenin-Nukleotiden vor einem schnellen Abbau geschützt. Die Introns werden noch während der Transkription von einer mit der RNA-Polymerase II assoziierten molekularen Schneidemaschine, dem Spleißosom, entfernt. Die von der RNA-Polymerase II abgelesenen microRNAs werden - nachdem sie sich zu kurzen Doppelsträngen zusammengelagert haben – in mehreren Schritten von einem als Dicer bezeichneten Enzym auf die richtige Größe angepasst. Das Dicer-Enzym wird während der Transkription zusammen mit anderen Hilfsproteinen von einem auf der Methyl-Guanosin-Kappe aufsitzenden Komplex rekrutiert [1, 2]. Weil Gene gerichtet abgelesen werden, muss die RNA-Polymerase II den Startpunkt identifizieren können. Dieser ist an einigen konservierten Sequenzabschnitten zu erkennen und wird als Promotor bezeichnet. Die RNA-Polymerase II bindet an diese konservierten Elemente und wird dabei von den Transkriptionsfaktoren TFII A, D, E, F und H und einem Komplex namens Mediator unterstützt. Der Mediator-Komplex stabilisiert am Promotor weitere positiv und negativ wirkende Transkriptionsfaktoren.
Die größte Untereinheit der RNA-Polymerase II hat einen langen C-terminalen Schwanz, der aus sich wiederholenden Blöcken von sieben Aminosäuren besteht. Man bezeichnet diese Struktur auch als die C-terminale Domäne der RNA-Polymerase II, abgekürzt RNAPII-CTD. Jeder dieser konservierten Blöcke, bestehend aus sieben Aminosäuren, trägt die Sequenz Y1S2T3P4S5P6S7, wobei Y für Tyrosin steht, S für Serin, T für Threonin und P für Prolin. Die Zahlen geben die Position der Aminosäure im Heptapetid an. Nachdem die RNA-Polymerase II einen Initiationskomplex auf dem Promotor ausgebildet hat, werden durch das selektive Anhängen von Phosphatgruppen an die drei Serine des Heptapeptids die weiteren Schritte eingeleitet [3].
Was wissen wir über die Transkription in Pflanzen?
Untersuchungen haben nun gezeigt, dass der Transkriptionsfaktor TFIIH bei Arabidopsis mit drei sogenannten Typ-D Cyclin H-abhängigen Proteinkinasen assoziiert ist. Die entsprechenden Abkürzungen sind CDKD;1 CDKD;2 und CDKD;3. Diese Kinasen koppeln eine Phosphatgruppe an das Serin in Position 5 und sorgen auf diese Weise dafür, dass die RNA-Polymerase II mit der Transkription beginnt. Sobald diese Markierung angebracht ist, werden weitere Proteine für den Transkriptionsprozess rekrutiert, und zwar solche, die der mRNA eine Kappe aus Methyl-Guanosin aufsetzen, zum Spleißosom gehören und die Anbindung des Dicer-Komplexes erleichtern, der regulatorische microRNAs passgenau spaltet [1, 3]. Eine weitere TFIIH-Kinase phosporyliert das Serin in Position 7. Dieses Enzym heißt CDKF;1. Nach unseren Erkenntnissen sorgt diese Markierung dafür, dass diejenigen Gene, die für die Herstellung der kleinen regulatorischen RNAs gebraucht werden, koordiniert abgelesen und dass die neu gebildeten Vorläufer der kleinen Ribonukleinsäuren über das Anhängen einer Poly-Adenin-Kette stabilisiert und geschützt werden [4]. Die Kinase, die das Serin 7 phosphoryliert, kann auch die CDKD-Kinasen und die CDKC-Enzyme aktivieren, die Serin 2 mit einem Phosphatrest beladen. Dadurch werden weitere Schritte der Transkription angestoßen. Die Serin 7-Kinase rekrutiert somit wichtige Faktoren, die für die Verlängerung, die Prozessierung und die Polyadenylierung der neu gebildeten Ribonukleinsäuren zuständig sind (Abb. 1).
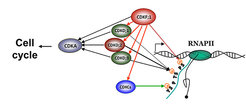
Abb. 1: CDKF;1 ist eine übergeordnete Kinase. Sie phosphoryliert das Serin 7 im repetitiven Heptapetid der RNA-Polymerase II und aktiviert diejenigen Kinasen, die das Serin 5 und Serin 2 mit einem Phosphatrest beladen. Diese Enzyme wirken auch auf die CDKA Kinase, die für den Zellzyklus (cell cycle) wichtig ist. Dadurch gibt es eine direkte Verknüpfung zwischen Transkription und Zellteilung.
Wie Transkription und Zellzyklus miteinander verknüpft sind
Neben ihren Aufgaben bei der Transkription aktivieren die beschriebenen Enzyme auch die CDKA-Kinase. Diese Kinase ist ein wichtiger Impulsgeber für den Zellzyklus. Deshalb spielen die CDKF;1 und die drei CDKD-Kinasen auch eine wichtige Rolle bei der Koordination von Zellzyklus und Transkription. Hemmt man zum Beispiel CDKF;1, wird die Zellteilung und Zellstreckung gehemmt, sodass derartige Pflanzen extrem zwergwüchsig sind (Abb. 2A). Eine Mutante, bei der das Serin 7 kaum noch phosphoryliert werden kann, produziert entsprechend wenig micro- und andere silencing siRNAs, die wichtige entwicklungs- oder hormonabhängige Stoffwechselwege kontrollieren (Abb. 2C). Weil wiederum die Phosphorylierung am Serin 7 ausfällt, geht auch die Phosphorylierung am Serin 5 zurück, was die Transkription weiter beeinträchtigt. Da die drei CDKD-Kinasen überlappende Funktionen wahrnehmen, führt ihre schrittweise Inaktivierung zu einer graduellen Hemmung des Wachstums (Abb. 2B). Werden alle drei Kinasen gleichzeitig gehemmt, ist die Pflanze nicht mehr lebensfähig. Weil bei einer partiellen Hemmung der Kinasen auch das Serin in Position 5 weniger phosphoryliert wird, haben die entstehenden Ribonukleinsäuren auch seltener eine schützende Kappe aus Methyl-Guanosin und werden nur noch vereinzelt durch Dicer prozessiert. Bei einer partiellen Hemmung sammeln sich somit viele unfertige Vorläufermoleküle der microRNAs an.

Abb. 2: Durch Inaktivierung von CDKF;1 und CDKD-Kinasen wird der Zellzyklus gehemmt und die Menge an microRNA geht zurück. (A) Die Inaktivierung von CDKF;1 führt zu Zwergwuchs. (C) Die Menge der kleinen RNAs ist reduziert. (B) Eine graduelle Hemmung der CDKD-Kinasen führt zu einer graduellen Wachstumshemmung und (D) zu weniger Pflanzentumoren nach einer Infektion mit Agrobacterium tumefaciens.
TFIIH Kinasen steuern Tumorbildung in Pflanzen
Die Daten zeigen, dass die Koordination zwischen Transkription und Zellteilung bei Arabidposis auf zwei Ebenen stattfindet: auf der Ebene der CDKF1-Kinase und der drei CDKD-Kinasen. Bei Pflanzen entstehen Tumore nur sehr selten spontan. Es ist deshalb besonders bemerkenswert, dass die Hemmung der CDKF;1- und CDKD;2-Kinasen dazu führt, dass Agrobakterien weniger Tumore in Arabidopsis erzeugen (Abb. 2D). Wir haben kürzlich gefunden, dass CDKF;1 ein von Agrobacterium produziertes Protein phosphoryliert, mit dessen Hilfe Tumor induzierende Gene in die Pflanzenzellkerne eingebaut werden. Agrobacterium greift also auf diese wichtigen Kinasen zurück, was nicht wundert, denn der Mikroorganismus hat das Ziel, nach Infektion der Pflanzenzelle den Zellzyklus einzuschalten, um nachfolgend im entstehenden Tumorgewebe zu leben und sich fortzupflanzen.

